蛋白质组学的一个主要目标是提供能够诊断疾病和监测治疗的检测方法。这些检测方法需要对单个蛋白质具有灵敏度和特异性,并且在大多数情况下,对同一样品中的一种以上的蛋白质进行量化。
目前用于蛋白质组学检测的两种主要技术是基于质谱和亲和分子组合,如抗体。在本文的第一部分,描述了基于这些技术的最灵敏的现有检测方法,并与ELISA的金标准进行比较。
分析灵敏度被定义并与检测限相关,分析特异性被定义并显示取决于分子校对步骤,类似于那些在需要高保真度时应用于生命系统的步骤。报告显示,目前无论是质谱法还是亲和分子小组都没有提供多重检测所需的灵敏度和特异性的必要组合。
在本文的第二部分,描述了越来越多的使用额外校对步骤来结合灵敏度和特异性的检测方法。这些包括基于邻近连接和慢脱速率修饰适体的测定。
最后,本文考虑了在不久的将来可能出现的改进,并得出结论:进一步开发包含先进校对步骤的蛋白质组学检测方法最有可能提供必要的灵敏度和特异性的组合,而不会产生高昂的开发成本。
01
简介
蛋白质组学是对健康和疾病中蛋白质的大规模研究。为了研究的目的,生物体的蛋白质通常被划分为基于不同组织或细胞的蛋白质组。然后在实验中研究这些蛋白质组中的蛋白质,实验范围从旨在识别大量无偏见的蛋白质组的发现蛋白质组学,到旨在量化预选的蛋白质组的检测。
从发现蛋白质组学到检测,有一个趋势,即从高含量(大量的蛋白质被识别)和低灵敏度,到低含量和高灵敏度,本文将讨论这一趋势中的高灵敏度的一端。
血浆蛋白质组是本文中描述的蛋白质组学检测的主要目标,因为它的可及性,也因为其他蛋白质组脱落到血浆中的蛋白质被认为可以诊断身体其他部位的健康状况,但这导致的复杂性带来了巨大的挑战。
血浆可能包含大多数人类蛋白质以及来自其他来源的蛋白质,如细菌、病毒和真菌[1,2]。这些蛋白质的大部分(99%的质量)是以22种高丰度蛋白质的形式存在,就血清白蛋白而言,其浓度高达10 mM(50 mg/ml),但其他蛋白质,包括一些经常需要测定的蛋白质,其浓度要低得多。
最常引用的浓度范围是从血清白蛋白到IL-6(目前诊断检测中含量最少的蛋白质之一,在健康人中的浓度约为100 fM)的十个数量级范围,但许多可能对早期疾病有诊断作用的蛋白质可能以更低的浓度存在。
大约22,000个非冗余蛋白质(基因产品)的基本数量由于存在许多变体而得到扩展,这些变体可能具有显著的分子相似性,但由于翻译后修饰而具有不同的功能。大多数蛋白质在生化网络和途径中发挥作用,通常是作为多分子复合体的组成部分。
因此,一个蛋白质的活性不仅取决于其自身的浓度,而且还取决于与之相互作用或抑制的其他蛋白质的浓度。
一个有据可查的例子是细胞因子[3,4]。这个小信使蛋白家族参与了多种生化过程,如细胞生长、细胞分化、组织修复和重塑,以及免疫反应的调节。它们经常与其他细胞因子一起发挥其作用,因此关于单一细胞因子的信息不太可能揭示病人的健康状况。
因此,蛋白质组学检测必须能够量化可能以低浓度存在于样品中的多种蛋白质,而不受到可能以更高浓度存在的其他蛋白质的干扰。
成功实现这一目标的检测方法被称为灵敏度和特异性,其中灵敏度是衡量检测方法在检测低浓度目标蛋白方面的成功程度,特异性是衡量它在区分目标蛋白和样品中存在的其他蛋白方面的成功程度(这些灵敏度和特异性的定义不应该与诊断灵敏度和诊断特异性相混淆)[5]。
一些蛋白质的浓度在刺激下可能会有多达四个数量级的变化,因此任何检测的另一个重要特征是动态范围,这是衡量目标蛋白质能够被准确量化的浓度范围。
目前用于检测蛋白质组的两种主要技术是质谱法(MS)和多重免疫检测。本文比较了基于这些技术的检测方法的优点和缺点,并研究了它们在多大程度上能够提供所需的灵敏度和特异性的组合。
02
质谱蛋白质组学
许多版本的MS已经被用于蛋白质组学[6-8],但大多数涉及将蛋白质消化成肽,然后用HPLC、气相色谱或毛细管电泳进行分馏。
分馏后的肽被送入质谱仪,质谱仪有三个主要部分:电离源、分析器和检测器。肽被电离并提取到分析器中,在那里根据它们的质量/电荷(m/z)比率进行分离和检测。随后,通过与肽数据库的比较,对原始蛋白质进行离线鉴定。
最简单的非目标版质谱可以在大约1小时的时间范围内识别数百种蛋白质,但许多蛋白质无法检测,因为它们的存在被更丰富的蛋白质所掩盖。因此,质谱的一个主要趋势是开发技术,通过减少其他蛋白质引起的背景信号来提高灵敏度。
一些技术通过减去样品中的干扰分子来减少背景,同时允许目标蛋白或肽流向检测器[9]。减法技术的例子有HPLC、凝胶电泳和免疫亲和力耗竭。
通过考虑一个具体的例子可以看出减法的效果。
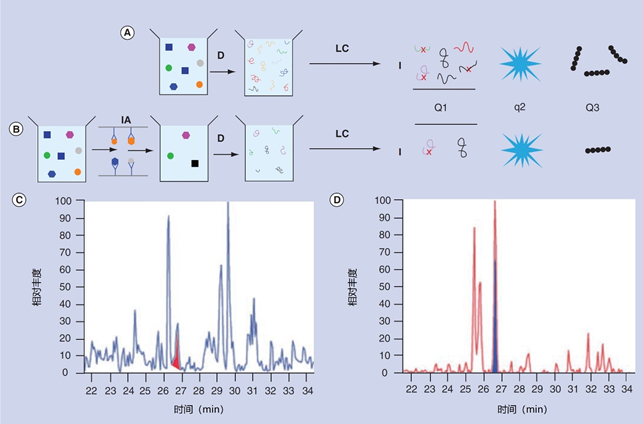
图1 | 消耗丰富的蛋白质对质谱分析的灵敏度的影响。
(A)全血浆的多重反应监测分析。
(B)对全血浆进行免疫亲和力耗尽丰富蛋白质的多重反应监测分析。
(C)无IA消耗的离子电流图,凝胶蛋白的峰值用红色阴影表示。
(D)用IA耗尽20种丰富的蛋白质的离子电流图,凝胶蛋白的峰值用蓝色阴影表示。
缩略语:D,样品的酶解;I,肽的电离;IA,免疫亲和;LC,液相色谱;Q1,分析仪1;Q2,碰撞池;Q3,分析仪2。
MRM(多反应监测,也被称为选择性反应监测)版本的MS是在一个串联质谱仪中进行的,它由两个分析器组成,被一个碰撞池隔开,如图1A所示。来自多达100个消化的目标蛋白的特征离子在第一台分析器中被选择,并在碰撞池中被分割。
在检测前,这些碎片在第二台分析器中被分离,结果以总离子电流图的形式呈现,如图1C所示。
图1B显示了MRM的工作流程,其中有一个额外的子牵引步骤,即用免疫亲和柱结合血浆中20种丰富的蛋白质;相应的离子流图显示在图1D。对比图1C和D可以看出,由于额外的减法步骤减少了背景信号,对应于目标蛋白(凝胶蛋白)的峰相对更大、更明显。
用免疫亲和力减去丰富蛋白质的MRM的动态范围约为四个数量级,灵敏度约为10 ng/ml(相当于分子量为50 kDa的蛋白质的200 pM),这对于检测许多低丰度的蛋白质来说不够灵敏。
所有的减法技术都会稀释样品,也可能会非特异性地减去目标分子,因此,应用一种以上的减法技术并不一定能提高灵敏度。然而,最近在15 μl血清中检测到了低pmoL浓度的前列腺特异性抗原,其工作流程是先用高pH值的反相HPLC,再用低pH值的反相HPLC和MRM[10]。
这种策略(被称为PRISM)的一个缺点是,即使在低重复水平下,每周也只能检测50个样本。样品产量低是基于MS的蛋白质组学检测的一个共同特点;一般来说,在一定时间内可以检测的样品数量随着灵敏度的提高而减少。
替代减法技术的一种方法是通过从样品中提取目标分子来减少背景[11]。
提取方法包括那些提取整类蛋白质的方法,如凝集素亲和色谱法,以及基于抗体提取目标蛋白质或肽的方法。一般来说,萃取技术比减法技术有更高的灵敏度,因为它们浓缩了目标分子而不是稀释了它们,但在最灵敏的情况下,它们可以与基于亲和分子的蛋白质组学检测直接竞争。
03
ELISA,蛋白质检测的金标准
亲和分子与具有互补的形状和化学性质的目标物质结合。到目前为止,抗体一直是蛋白质组学中使用最广泛的亲和分子。抗体结合位点和相应的目标位点之间的平衡可以表示为:
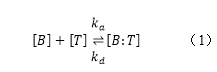
其中[B]和[T]是未被占用的抗体结合位点和未被结合的目标位点的摩尔率,[B:T]是被占用的结合位点的摩尔率。公式1中ka和kd是结合和解离的速率常数。结合和解离速率常数的比率被称为抗体的平衡常数或亲和力常数(Ka)。
高亲和力的抗体要么与目标物质结合得更快(高ka),要么与目标物质保持更长时间的结合(低kd),或者同时具有这两种特性。
基于抗体的蛋白质组学检测(免疫检测)可以分为两种形式,取决于结果是通过测量被占据的还是未被占据的结合位点来确定[12]。前者被称为试剂溢出型免疫检测,后者被称为试剂限制型免疫检测。
试剂限制型免疫检测一般用于检测那些太小而不能容纳一个以上的抗体结合位点的物质。免疫竞争法检测是试剂限制性免疫检测的例子。
试剂溢出型免疫检测通常是用两个与同一目标物质上的不同结合位点(表位)结合的抗体来进行的,免疫夹心法是试剂溢出型免疫检测的例子。
这两种形式都可以用一种酶作为标签,但是当人们把ELISA称为蛋白质组学检测中灵敏度的金标准时,他们说的是试剂溢出型的免疫夹心法。
ELISA是指酶联免疫吸附试验,但使用其他标签的免疫夹心法检测,如镧系螯合物[13]、吖啶酯[14]和钌系螯合物[15],也有类似的灵敏度。这些单一目标物质的免疫检测通常是在96孔或384孔的多孔板中进行的,图2A显示了一个简单的ELISA的主要步骤。
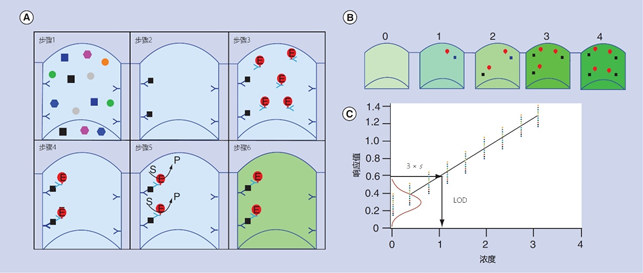
图2 | ELISA,蛋白质检测的金标准
(A)酶联免疫吸附夹心法的关键步骤(解释见正文)。
(B)酶产物的数量与结合的目标物质的数量成正比,而目标物质的数量又与原始样品中目标物质的数量成正比。
(C)可以根据一系列含有已知浓度的目标物质的校准溶液的反应(可检测的产物量)来构建校准图。红色的高斯曲线是基于零校准液的标准偏差(s)。LOD可以通过在零号校准液的3 × s处水平投影直到与反应线相交来确定,然后将交点向下垂直投影到X轴上。在这个例子中,LOD是1.16。
缩略语:E,酶标;LOD,检测限;P,酶催化反应的产物;S,酶底物。
在步骤1中,样品与固定在板孔中的捕获抗体进行孵化。在步骤2中,样品被取出,孔被清洗。在步骤3中,将带有酶标记的检测器抗体加入到孔中,并进行第二次孵化。在步骤4中,去除未结合的检测器抗体,并清洗该孔。在步骤5中,加入底物并进行第三次孵化,其中酶催化底物转化为可检测的产物,如步骤6中所示。
如图2B所示,产物的数量与原始样品中目标物质的数量成正比。通常情况下,ELISA需要1至2小时才能完成,其动态范围为三个数量级。
图1B和图2A之间的比较说明了减法和萃取法之间的关键区别:在减法(图1B)中,不是目标物质的物质被从样品中提取出来,而目标物质继续流向检测器,但在萃取法(图2A)中,目标物质被提取出来,样品的其余部分被丢弃了。
04
抗体微阵列与ELISA的灵敏度
多重一词有多种含义,但在本文中,除非另有解释,它是指在相同的未分割的样品体积中检测一种以上的目标物质。在多重免疫检测的早期,人们普遍认为高重数(高含量)的测定将通过扩大现有的单分子方法来开发。
多重免疫分析最常见的方法是将不同目标物质的抗体限制在被称为微阵列的微米大小的矩形网格中的已知位置[16-19]。
第一个抗体微阵列与DNA微阵列差不多同时出现。从那时起,DNA微阵列的发展取得了重大进展,并取得了相当大的商业成功。现在,商业化的DNA芯片已经可以用于突变分析、表达分析和SNP检测,年销售额达数亿美元。
在开发抗体芯片方面的技术进步则更为有限。多重免疫夹心微阵列的发展(图3A)被开发同一目标物质上不同表位的配对抗体的成本所阻碍,这种成本随着测定内容的增加而成倍增加。
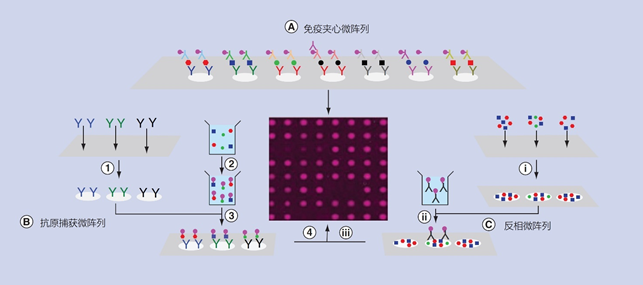
图3 | 用于检测蛋白质的三种微阵列类型。
(A)已完成的免疫夹心微阵列:工作流程与ELISA相似,但标签必须产生一个不扩散离开其在网格中的位置的信号。
(B)开发抗原捕获微阵列的主要步骤:(1)在平面基质上打印抗体微阵列;(2)标记样品中的分子;(3)将标记的样品与微阵列孵化;(4)获取完整微阵列的图像。
(C)开发反相微阵列的主要步骤:(ⅰ)在平面基质上打印样品;(ⅱ)将特定目标物质的标记抗体与微阵列孵化;(ⅲ)获得完整微阵列的图像。
开发单一表位的抗体的成本要低得多,因此大多数的多重免疫微阵列是基于每个目标物质的一个抗体。
这些单抗体免疫微阵列的两个主要形式是抗原捕获微阵列(图3B),其中样品被标记,并用抗体微阵列进行检测;以及反相微阵列(图3C),其中固定肽、抗原、蛋白质、细胞或组织样品的微阵列用标记的抗体进行检测。在大多数情况下,多重免疫微阵列是在平面基质上进行的,但也可以用编码颗粒的悬浮微阵列进行[20,21]。
一种检测方法的分析灵敏度是衡量其区分同一物质的不同浓度的能力,但通常这一定义被更狭义地解释为区分含有和不含有该物质的样品。这种狭义的解释通常被称为测定的检测限(LOD)。更正式的定义是,LOD是指目标物质不被检测到的概率α(阴性结果)的浓度[22]。
最广泛选择的α值是0.015,它相当于零校准器(已知没有目标物质的样品)标准偏差(s)的三倍。如果α的值为0.015,那么LOD可以通过在Y轴上水平投影零校准器的m + 3 × s(其中m是平均反应)的点,直到它与最适合的剂量-反应线相交,然后将交点向下投影到X轴,如图2C所示。
LOD的可靠性取决于它所基于的测量数量,因此用于确定LOD的重复数(n)也应该被报告;对于广泛使用的95%的置信区间来说,至少n = 6是可以接受的。在严格开发的免疫夹心微阵列中,s的大小主要取决于非特异性结合的数量和随着测定的进行而积累的误差。
如果假设标签具有无限的特异性活性,并对非特异性结合量和误差进行假设,就可以用公式3来估计试剂残留免疫分析的LOD:
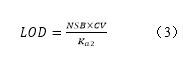
其中非特异性背景信号(NSB)是非特异性结合量占标记的检测器抗体总浓度的一部分,CV是与零校准物相关的相对误差(s/m),Ka2是检测器抗体的平衡常数或其等价物[23]。注意NSB在这里是决定灵敏度的关键因素,就像在MS中一样。
图4显示了如果假定NSB和CV的值为0.001和0.01,免疫夹心微阵列的灵敏度如何取决于Ka2。0.001的非特异性结合和0.01的相对误差是在实践中可以实现的低端,而良好的分析级蛋白质单克隆抗体的Ka值在1×10^10左右。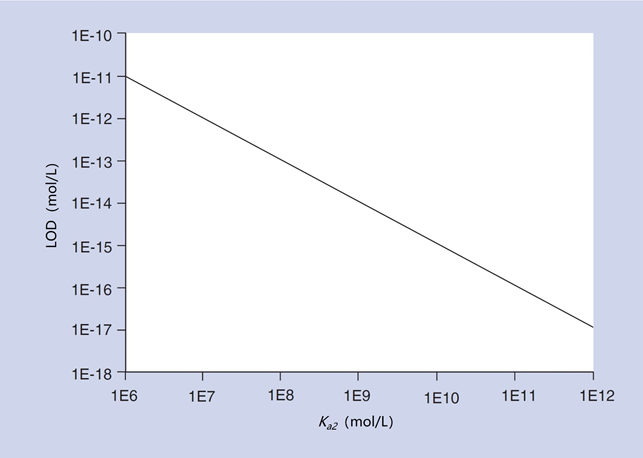
图4 | 免疫夹心法检测的检测限取决于检测器抗体对结合目标物质的亲和力常数。
缩略语:LOD,检测限。
因此,图4预测免疫夹心微阵列的灵敏度将集中在1×10^-15 mol/L的浓度上。由于TSH在临床上的重要性,很多人都致力于开发TSH的免疫夹心微阵列。对商业来源的TSH免疫微阵列的审查发现,其灵敏度在0.005 ~ 0.1 mIU/L之间[24]。
如果假设人类TSH的活性为5 IU/mg,MW为28 kDa,这相当于灵敏度在35 ~ 714 × 10^-15 mol/L之间。如图5所示,对免疫夹心微阵列的报告LODs的广泛调查表明,它们集中在50 × 10^-15 mol/L的标准浓度上。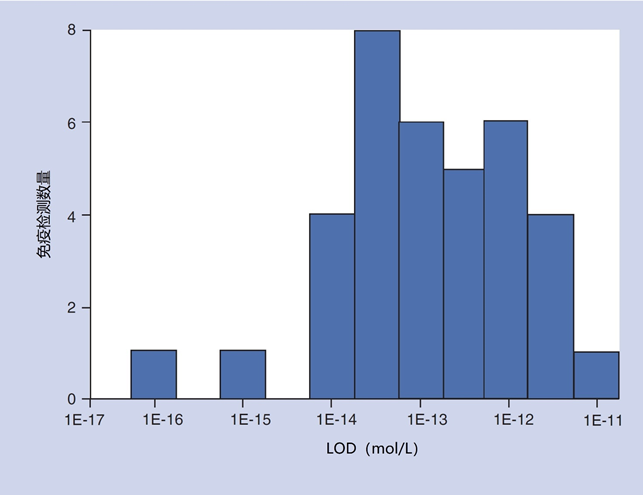
图5 | 免疫夹心法(ELISA和时间分辨荧光)的检测限报告。
缩略语:LOD,检测限。
这些对灵敏度的估计适用于试剂溢出型检测,在这种检测中测量被占领的结合点,如免疫夹心法检测。测量未被占据的结合位点的亲和力方法(如免疫竞争法检测)的灵敏度较低,因为它们涉及到在大背景下检测一个小信号。
理论模型表明,这种类型的检测方法的灵敏度可以通过亲和力常数(Kd)的倒数乘以相对误差(即CV × Kd)来估计[25]。因此,使用Kd = 1 × 10^-10和CV = 0.01抗体的免疫竞争法检测LOD将是1×10^-12 mol/L。
在实践中,免疫竞争法检测主要用于检测低分子量的分子,对于这些分子,抗体的Ka值通常低于1 × 10^10,因此灵敏度也相应较低。
经常有人声称,抗体捕获微阵列免疫检测的LODs在pM-fM范围内具有灵敏度(可与ELISA相媲美)[26-30],但是没有统计学上的可靠结果来支持这一说法。
在其中一项较好的研究中,抗原捕获微阵列是通过将8种补体蛋白的单链抗体结合位点点缀在黑色聚合物微阵列玻片上而制备的[31]。微阵列与用生物素标记的血清和血浆样品进行孵化,然后用荧光链霉菌素标记。
通过减去每个点的局部荧光来确定反应,以补偿背景信号的局部变化,并使用一个内部对照来使不同微阵列的反应正常化。每个蛋白质的两个最高和最低的反应被排除,LODs是基于其余四个重复(n = 4)。
八种补体蛋白的LODs(基于2 × s的零校准物)在0.35 ~ 530 pM范围内(八种目标细胞因子的平均LOD=10^7 pM),这比ELISA的灵敏度低三个数量级,但比MRM版本的MS更灵敏。
公式3显示,决定灵敏度的一个关键因素是背景信号的大小。在免疫夹心微阵列中,背景信号主要是由于非特异性结合的标签造成的,这取决于所用的标记抗体的浓度(图2A的步骤3)。在抗原捕获微阵列中,样品中的所有分子都被标记,因此它们都有可能导致非特异性结合。
因此,公式3中的NSB对于抗原捕获微阵列来说不可避免地要高得多,因此它们的灵敏度低于ELISA和其他设计良好的免疫夹心微阵列也就不足为奇。反相微阵列可能比抗原捕获微阵列更灵敏,因为NSB取决于标记的抗体的浓度,而不是样品中所有标记的分子。
05
特异性的来源,分子校对步骤
一个抗体的分析特异性是衡量它在指定的目标物质和样品中存在的其他物质之间区分的有效程度。与抗体结合的其他物质被称为交叉反应物。特异性取决于交叉反应物的相对浓度和亲和力。
高浓度的交叉反应物可以扭曲用抗体检测低丰度目标物质的结果,即使目标物质的Ka比交叉反应物的亲和力常数(Kac)高很多。缺乏特异性仍然是阻碍发展多重免疫微阵列的主要障碍之一。
免疫检测中的各个步骤可以被看作是一系列的分子校对步骤,导致误差分数(交叉反应物与目标物质的比率)的逐步减少。
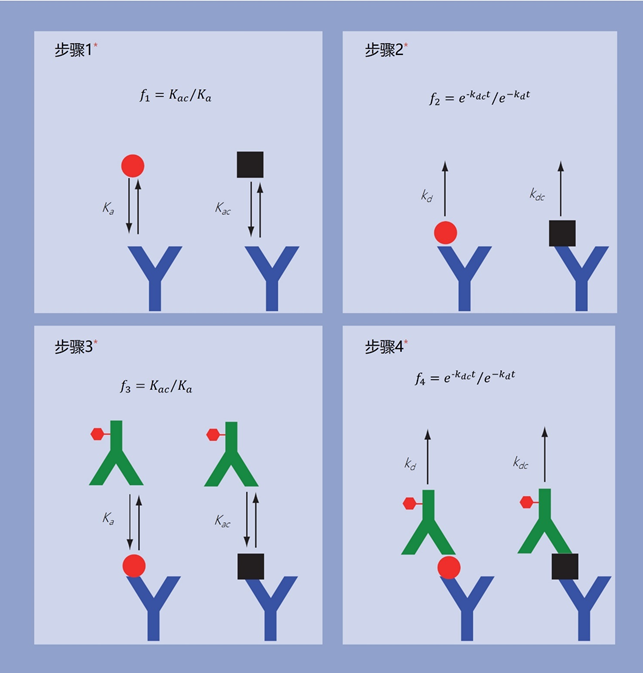
图6 | 免疫夹心法的四个阶段被视为校对步骤,其中错误分数(预定的目标分子[红色圆圈]与交叉反应物质[黑色方块]的比率)被连续削弱。插图中的公式显示了如果只有一种交叉反应物质,每个步骤的误差分数将如何计算。
图示:每个红色星号对应于一个校对步骤。
图6显示了双位点免疫检测中每个步骤的误差分数(f)。图6中显示的f的插入公式是基于以下假设:步骤1和3达到平衡;步骤2和4的持续时间为t秒;步骤1中抗体的浓度小于(0.05/Ka);步骤1中目标物质和交叉反应物质的浓度相同,且小于(0.001/Ka)。免疫检测的总体误差分数(ft)为:
ft=f1×f2×f3×f4
表1显示了如果Ka = 10^10,Kac = 10^8,kd = 10^-5/s,kdc = 10^-3/s和t = 300 s,免疫检测中每一步的f和f值。根据这些值,四步后的累积误差分数ft比两步后的ft小两个数量级。这种类型的计算表明为什么两步抗原捕获微阵列的特异性低于四步免疫夹心法检测。
表1 | 基于Ka 10^10,Kac 10^8,kd 10^-5/s,kdc 10^-3/s和t 300 s的简单免疫夹心法检测中每个步骤的误差分数(f)和累积误差分数(ft)

尽管灵敏度和特异性较低,但由数百种抗体组成的抗原捕获微阵列在研究中被广泛使用。一个常见的应用是生物标志物的发现,这涉及到用抗体微阵列比较健康(对照)和疾病(病例)样本的结果[32-36]。
通常情况下,这些微阵列是用商业来源的抗体制备的,用于命名目标物质。Loch等人使用由320个整体单克隆和多克隆抗体组成的抗原捕获微阵列来比较卵巢癌病例和对照组的血清样品[36]。
一些目标物质用一种以上的抗体进行了检测。例如,蛋白质CA125(一种已知的卵巢癌生物标志物)被用八种不同的抗体检测,这些抗体给出的不能区分癌症患者和对照组的概率分数(相同队列大小的P值)从0.003到0.171不等,错误发现率(FDRs)从0.214到0.433(即每100次检测,错误阳性的数量在21到43之间,取决于抗体的选择)。
这些巨大的变化表明,用抗原捕获微阵列获得的结果可能在很大程度上取决于抗体的选择,而不是病例和对照之间的任何实际差异。
抗原捕获微阵列的使用是基于抗体对单一目标物质具有特异性的假设,但是十多年前,人们发现115个微阵列的抗体中只有20%对其目标物质产生了定量的正确结果[37]。
最近,当用酵母蛋白的反相微阵列研究11种商业来源的抗体的特异性时,发现有5种抗体对其目标物具有特异性,5种抗体与多种抗原发生交叉反应,1种抗体与1000多种不同的抗原结合[38]。
在另一项研究中,用重组蛋白的反相微阵列对六种商业来源的抗体进行了检测,发现有两种抗体是特异性的,但有四种抗体与多达12种其他蛋白表现出强烈的交叉反应[39]。
这些结果表明,许多单个抗体的特异性不足以识别其指定的目标物质,并有助于解释为什么很少有生物标志物研究被转化为临床试验。
06
MS与亲和力分子的结合,
灵敏度/特异性问题的解决方案
提高蛋白质组学检测的特异性的一种方法是将抗原捕获与质谱结合起来。这种组合被称为稳定同位素标准与抗肽抗体捕获(SISCAPA),被Whiteaker等人用来检测血浆中的9种蛋白质,如图7所示[40]。
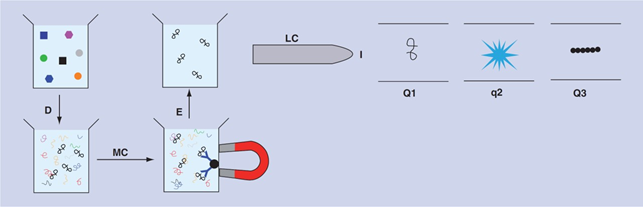
图7 | 用抗肽抗体捕获的稳定同位素标准,抗原捕获和质谱分析相结合。
缩略语:D,样品的酶解;E,肽与抗体的解离;I,肽的电离;LC,液相色谱;MC,与抗体结合的肽的磁捕获;Q1,分析仪1;Q2,碰撞池;Q3,分析仪2。
用胰蛋白酶消化样品(每种蛋白质检测10 μL血浆),然后用抗体和磁珠捕获每种目标蛋白质的一个肽。选择肽是因为它们是目标蛋白所特有的,并且在MS中表现良好,但不是因为它们是抗体的好目标。
从抗体中洗脱出来的多肽用液相色谱法进行分馏,并使用MRM方法进行MS检测。一开始有15种蛋白质被作为目标,但后来有6种因为在抗体捕获阶段回收率低(<7%)而被放弃。其余检测的特异性很高,因为MS被用作高保真校对步骤以及检测。
检测的动态范围为三到四个数量级,平均LOD(m + 3 × s)为88 pM,这与抗原捕获微阵列相似,但比ELISA低两到三个数量级。通过从更大体积(1 mL)的样品中提取多肽,LODs被降低到3 pM,但在许多情况下不会有这么大体积的样品。
尽管LODs与抗原捕获微阵列相当,但后者的多重版本具有不那么复杂的工作流程和更快的吞吐量,并且需要更小的样品量。
SISCAPA与ELISA的比较表明,由于在抗原捕获阶段多肽的不可重复的回收而导致的不准确,但这可能会被一个不那么复杂的工作流程所改善。
将抗体捕获的灵敏度和质谱的特异性结合起来,听起来像是对灵敏度/特异性问题的解决方案,但一个检测方法只有在其最薄弱的环节才是好的,而SISCAPA的薄弱环节是质谱的灵敏度。
因此,这一领域的进展将取决于质谱灵敏度的提高,但要与下面描述的基于亲和分子的多重检测竞争,必须弥补三到四个数量级的差距,这在不久的将来是不可能被跨越的。
07
系统产生更多亲和力分子
使用抗原捕获微阵列而不是免疫夹心微阵列的一个常见原因是没有针对同一目标物质的不同表位的匹配抗体对。对更广泛的廉价亲和试剂的需求是几个大型开发项目的主题。
人类蛋白质图谱项目已经开发了一个以基因为中心的管道,用于系统生成和验证亲和纯化的多克隆抗体[41,101]。这条流水线的起点是在计算机中识别与其他蛋白质同源性低的80 ~ 100个氨基酸表位标签。这些标签被表达为重组蛋白片段并用于在兔子体内培养抗体。
用相同的表位标签作为配体,通过亲和层析法纯化抗体,并用肽的反相微阵列进行验证。
如果一个抗体的大部分结合是针对正确目标的抗原,那么它就被认为是有效的。我们的目标是针对同一蛋白质上不重叠的表位产生至少两个验证的抗体。
当下,已经有超过34000个抗体被验证,并且新的抗体正在以每月492个的速度增加,但是这些数字是基于一个非常弱的验证形式。在更严格的Western Blot验证中,22,000个测试的抗体中只有531个抗体与预期的MW的单一条带结合[42]。
一些额外的条带可能是由于同一蛋白质的变体或复合物造成的,但是这一点还没有被证实。人类蛋白质图谱中发表的所有抗体都可以在市场上买到,但是到目前为止,还没有任何抗体被转化为有效的诊断测试。
亲和蛋白质组专注于使用高通量体外选择方法作为增加可用亲和试剂范围的手段[43,102]。全长抗体中只有一小部分参与了与目标分子的结合。这一部分可以通过噬菌体和核糖体展示等方法,在体外表达为与编码它的DNA相关的肽。
然后,通过对多肽组合库进行反复的亲和力捕获和编码DNA的扩增循环,就可以鉴定重组抗体。这个过程比在动物身上生产抗体的方法要快(几周而不是几个月),而且成本较低,因为它可以在小体积的溶液中进行,需要的基础设施较少。
也有可能为那些对动物宿主有毒或因与动物自身抗原相似而无法产生反应的目标生产重组抗体。体外方法还可以包括一些条件,以提高对目标分子的微小改变(如翻译后修饰)的识别能力,或减少对可能的竞争对手的交叉反应[44,45]。
在某些情况下,体外选择被用来提高重组抗体的Ka值,使其超过体内施加的大约10^10的上限[46,47],但提高亲和力并不一定能提高特异性。大多数重组抗体的亲和力可与动物体内产生的抗体相媲美。
08
包括更多校对步骤的
特异性基因分型检测法
缺乏特异性并不局限于基于抗体的检测。它对核酸检测的影响在单核苷酸多态性(SNP)的检测中最为明显,在这种检测中,有必要对仅有一个碱基差异的序列进行区分[48]。
明确的核酸化学允许在严格的条件下通过将核酸序列与固定的寡核苷酸探针微阵列进行杂交来克服这个问题,但每个SNP必须用多个探针进行检测,以区分完全匹配和不匹配的杂交。
例如,由Affymetrix基因分型微阵列鉴定的每个SNP要用40个不同的探针进行检测[49]。一个由四个探针组成的区块包括与SNP完全匹配的探针和三个与完全匹配的探针,除了在SNP的基因座上有一个不同的碱基;这些碱基位于探针序列的中间位置。
另外两个由8个探针组成的区块,所有这四个碱基都从序列的中间移到±1和±4个碱基。第四块20个探针由其他20个探针的反义版本组成。通过从完美匹配产生的更强烈的信号中减去交叉反应探针的反应来鉴定SNP。
蛋白质的化学成分比核酸更多样化,因此不能用抗体产生相当于基因分型的微阵列,但也可以通过执行一系列校对步骤而不是用多个探针检测来检测SNPs。在golden gate检测法[50]中,基因组DNA被生物素激活并附着在链霉菌素磁珠上。
在检测的步骤1,磁珠与等位基因特异性探针(ASP)和基因组特异性探针(LSP)一起孵化,前者与基因组目标序列杂交,其3′端与SNP的位置相匹配,后者与ASP的上游杂交,如图8所示。
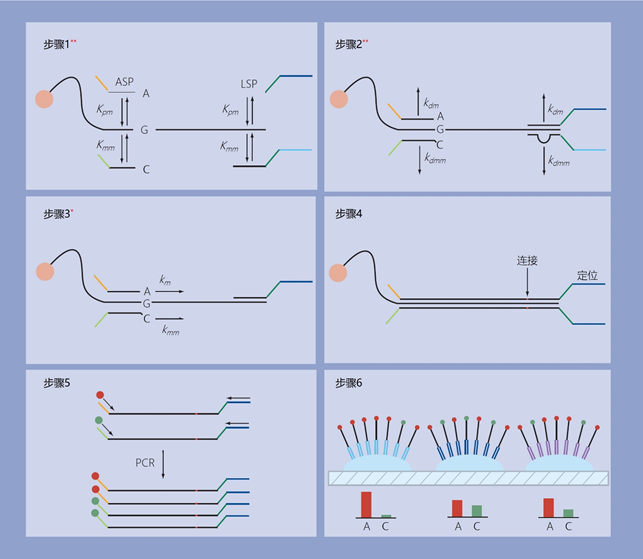
图8 | golden gate基因分型检测,主要校对步骤的位置和数量由插入的红色星号表示。
步骤1:ASP和LSP与附着在磁珠上的基因组目标序列进行竞争性杂交,这是两个校对步骤。
步骤2:严格清洗,优先分离不正确的杂交探针,这是两个校对步骤。
步骤3:用匹配的3′端碱基延长ASP,这是一个校对步骤。
步骤4:将延长的ASP与LSP连接。
步骤5:用荧光引物对连接的探针进行PCR。
步骤6:将PCR产物与光纤板中的探针微阵列进行杂交。
缩略语:ASP,等位基因特异性探针;LSP,基因座特异性探针。
在步骤2中,对珠子进行严格的洗涤,使不正确的杂交探针分离,在步骤3中,用一种对SNP基因座上的错配高度灵敏的聚合酶扩展ASPs。在步骤4中,用DNA连接酶将扩展的ASPs与LSPs连接起来,在步骤5中,使用ASPs和LSPs中的通用PCR位点扩增连接的探针。
在步骤6中,PCR产物与LSP中地址序列互补的寡核苷酸微阵列进行杂交。该检测的前三个步骤包含五个校对步骤。这些校对步骤中错误分数的累积衰减使得SNPs可以在不使用大量探针的情况下被鉴定。
如图6所示,前四个校对步骤与免疫夹心法检测中的校对步骤相当,但没有相当于第五个校对引物延伸步骤。这些校对步骤类似于赋予许多生物高特异性反应动力学校对方案中的步骤[51]。
在这些方案中,通过扩大所需结果的自由能变化和非特异性副反应的自由能变化之间的差距,总体误差部分被逐渐减弱。只要有高特异性的要求,生物系统就会采用这种方案,这一观察表明,基于亲和分子的蛋白质组学检测的特异性也可以通过增加校对步骤的数量和/或严格程度来提高。
09
通过增加校对步骤提高亲和
蛋白质组学的特异性
在免疫检测中增加校对步骤的一种方法是将其与MS使用的一种分馏技术相结合[52]。Wu等人使用尺寸排除色谱法将生物素标记的细胞裂解液分解为10到670 kDa的20个馏分。每个馏分都与300个连接到编码微珠的抗体进行孵化,然后用荧光链霉菌素标记与洗净的微珠结合的抗原,并通过流式细胞仪检测。
作者表示,一些蛋白质在只包含几千个细胞的样品中可以检测到,但由于没有LOD,所以不可能与其他方法比较灵敏度。只要交叉反应物质不与目标物质在同一馏分中洗脱,特异性肯定会提高,但灵敏度可能会随着样品稀释导致的馏分数量增加而降低。
免疫检测中最著名的附加校对步骤的例子是接近连接[53,54]。在接近连接免疫检测(PLA)中,抗体被连接到一个寡核苷酸接近探针上,该探针由一个靠近抗体的独特识别序列和一个普通连接序列组成。
当一个分子被夹在两个抗体之间时,探针被带到接近的地方,在那里与一个连接的寡核苷酸杂交在热力学上是有利的。
在一个多重检测中,抗体结合反应是在溶液中进行的,但在低温下过夜孵化是为了促进热力学上最有利的杂交。然后探针通过酶联接形成报告序列,该序列被扩增并通过qPCR检测。
这允许对24种蛋白质进行灵敏的多重免疫夹心法检测,而不需要进行大量的抗体选择和优化[55]。
这种无分离版本的PLA的主要缺点是接近探针的浓度必须低,以避免非特异性连接,因此检测的动态范围很短[56]。这一限制可以通过使用分离和洗涤步骤来克服,如图9所示,这样可以进行额外的校对步骤[57]。
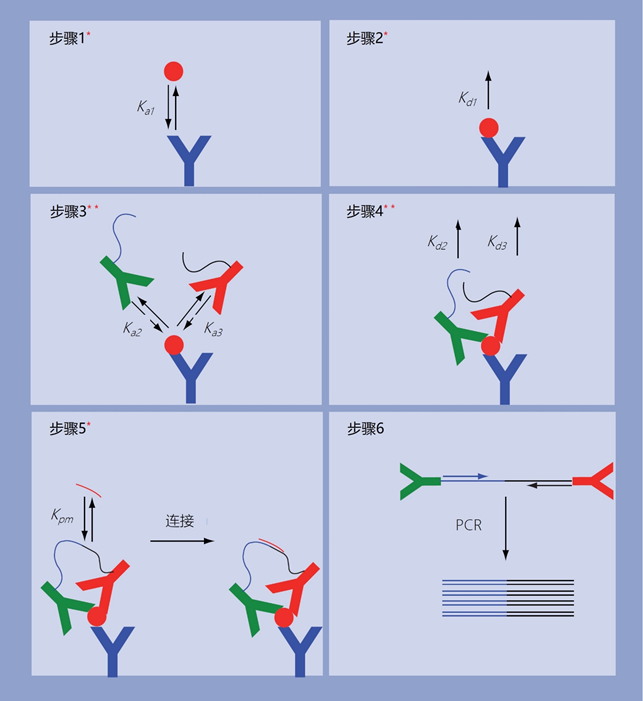
图9 | 在[57]中描述的接近连接免疫检测,主要校对步骤的位置和数量由插入的红色星号表示。
步骤1-4与免疫夹心法检测相似,只是通过使用两个检测器抗体来增加校对步骤的数量。检测器抗体用寡核苷酸标记,只有在步骤5中,如果它们的位置很近,才会连接在一起。在步骤6中,通过实时PCR检测已经连接在一起的寡核苷酸。
这个版本的PLA的平均LOD(m + 2 × s)为10 fM,与ELISA的最佳状态相当,而动态范围为5到6个数量级,效果更好。在图9所示的例子中,通过使用两个检测器抗体,特异性得到进一步加强。还有一种原位版的PLA,用于细胞和组织中的单个蛋白质和蛋白质-蛋白质复合物,连接后形成一个圆形模板,通过局部滚动圈扩增检测[58]。
第一个关于PLA的报告部分是基于两个与凝血酶不同表位结合的DNA适配体[59]。适配体是单链核酸,具有与抗体相当的识别特性,因为它们可以折叠成复杂的三维形状,与广泛的目标实体特异性结合[60,61]。
它们是通过筛选10^12-10^16个寡核苷酸的组合库来获得与所选目标分子结合的图案。筛选过程被称为指数富集配体的系统进化(SELEX),包括体外选择和PCR扩增的反复循环。
在选择步骤中,单链寡核苷酸竞争目标物质上的表位,而在扩增步骤中,寡核苷酸池被富集了结合的序列。然后,富集的池子被送入下一个选择和扩增周期。通常情况下,要进行5到20个循环,然后通过克隆和测序鉴定剩余池中的适配体。
尽管有很多优点,但适配体还没有被广泛用于蛋白质组学检测,因为一般来说它们的亲和力比抗体低(因此灵敏度也低),如图10所示。
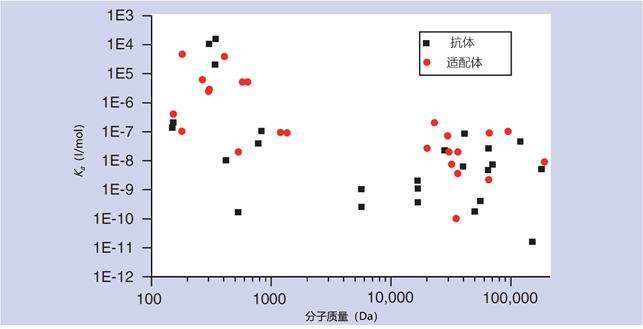
图10 | 抗体和DNA诱导体亲和力的比较,显示了它们如何取决于目标物质的分子量。当目标物质的分子量较低时,DNA适配体的亲和力与抗体相当,但一般来说,对高分子量物质的亲和力不如抗体。
另一个问题是,对于大多数蛋白质来说,所有已经确定的合剂都与相同的表位结合,因此无法获得夹心法检测中可能的额外校对步骤[62]。像图6所示的校对模型的一个优点是,它们揭示了哪些步骤最适合改进。
表1中的模拟值表明,解离步骤(步骤2和4)比结合步骤(步骤1和4)在减少错误分数方面的效果要差。最近,Gold等人引入了一类新的合剂,称为慢速分离率修饰的合剂(SOMAmers),这些合剂因其与目标分子的缓慢解离率(低kd值)而被选中[63,64]。
这使得亲和力测定中的洗涤步骤可以在更严格的条件下进行,从而导致非特异性结合的诱导剂的优先解离。这就弥补了第二个合拍体所提供的额外校对步骤。基于SOMAmers的多重检测,其平均LOD为300 fM(m + 3.2 × s),动态范围为7个数量级,已被用于识别各种医疗条件的生物标志物,包括肺癌和慢性肾病[63,65]。
10
蛋白质组学检测的现状和方向
正如本文标题所示,需要结合灵敏度和特异性的蛋白质组学检测方法。基于质谱和抗原捕获(SISCAPA)的检测方法具有高度的特异性,但它们的灵敏度不足以检测大多数低丰度的蛋白质。
免疫夹心法检测的灵敏度足以检测许多这样的蛋白质,但由于识别足够特异的匹配抗体对的成本很高,所以多重测定仅限于高价值的目标物质。
解决灵敏度/特异性问题的一个办法是提高SISCAPA的灵敏度,但灵敏度的差距很大,即使它可以被弥补,也很难看到基于MS的检测如何能够在资源丰富的实验室以外的地方部署。
随着蛋白质组学检测的目的从发现转向健康和保健,对可以在资源有限的地方部署的廉价蛋白质组学检测的需求将增加[66]。灵敏度/特异性问题的另一个解决方案是找到提高基于亲和力的多重检测的特异性的方法,而不产生高昂的开发成本。
基于抗体的多重检测可以部署在资源有限的地方[67,68],但对于大多数目标物质来说,使其发挥作用所需的抗体是不可用的。一些项目正在扩大可用的亲和力试剂的范围,但在开发多重免疫检测时产生的大部分成本来自于选择没有交叉反应的抗体,而不是抗体本身的产生。
因此,只有开发出成本较低的将亲和力试剂纳入多重检测的方法,扩大亲和力试剂的范围才能导致蛋白质组学的进展。PLA是这个方向上的一个重要进展,因为它降低了将抗体纳入多重检测的成本,但它需要匹配的一对甚至三对抗体[57]。
PLA最初是用诱导剂[59]演示的,但由于到目前为止只发现了一对经过验证的匹配的诱导剂(用于凝血酶),所以排除了使用这些亲和试剂的进一步工作[62]。这是不幸的,因为PLA与诱导体的组合比抗体更容易实现,并且具有更高的校对潜力。
DNA适配体属于一组分子,是抗体的廉价替代品。这些替代品包括通过噬菌体和核糖体展示技术分离出来的多肽适配体[69],但DNA适配体作为最简单和最经济的生产方式脱颖而出。
Gold等人成功地开发了上述高重数的多重检测方法,原因之一是他们的SOMAmers具有类似氨基酸的功能团[63,70]。这种化学反应是由Eaton等人开发的[71],并构成了一种方法的一部分,这种方法正朝着与酶促扩增相适应的类似肽的混合分子的方向发展[72]。
为什么只发现了一对匹配的适配体,一个建议是,与基于氨基酸的抗体结合位点相比,PCR可扩增的核苷酸的低化学多样性通常排除了匹配对[71,73]。引入更多的与PCR兼容的核苷酸化学成分开辟了新的设计空间,应该更容易识别除凝血酶以外的蛋白质的匹配对合剂。
这将与基于分子生物学工具的高级校对步骤的多重蛋白质检测相兼容[74]。基于肽类核酸诱导体和更多校对步骤的多重蛋白质组学将与目前的共识有很大的不同,但希望通过强调现有蛋白质组学检测中存在的问题,本文将为这一方向的进一步发展提供动力。
11
未来展望
对灵敏和特异的蛋白质组学检测的需求是MS或传统的抗体微阵列所不能满足的。质谱不够灵敏,传统的抗体微阵列要么不够特异,要么开发起来不划算。
蛋白质组学检测的理想灵敏度和基于MS的检测的实际灵敏度之间的差距非常大,在未来5年内不可能被弥补,但基于亲和分子的检测的特异性可以通过更好的校对来提高,而不会产生高昂的开发成本。
有两项发展特别显示了改进校对的优势:带有近似连接的抗体和SOMAmers。每种方法都有其特殊的优势。在未来五年内,预计结合这些优势的蛋白质组学检测方法将开始出现。

诊断科学编辑团队收集、整理和编撰,如需更多资讯,请关注公众号诊断科学(DiagnosticsScience)。
来源:诊断科学
声明:本平台注明来源的稿件均为转载,仅用于分享,不代表平台立场,如涉及版权等问题,请尽快联系我们,我们第一时间更正,谢谢!






