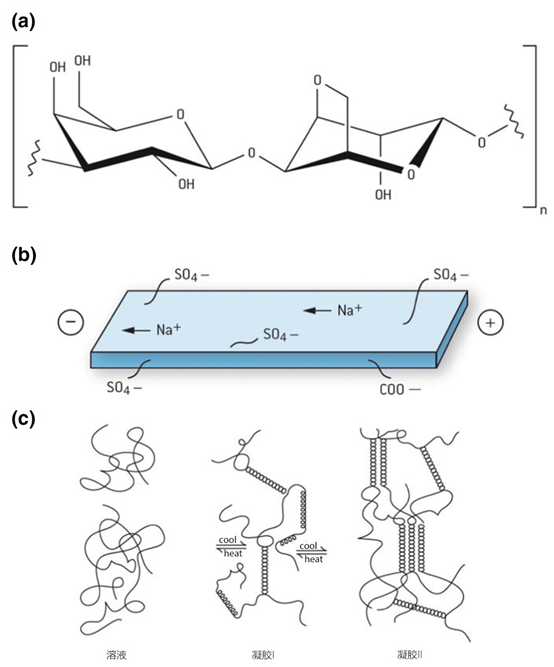
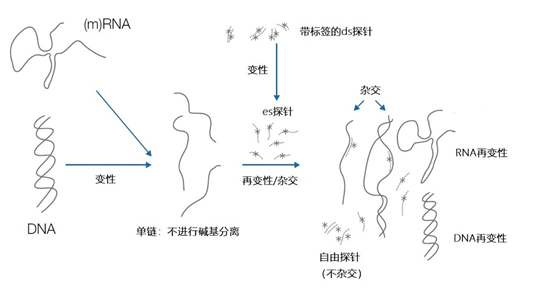
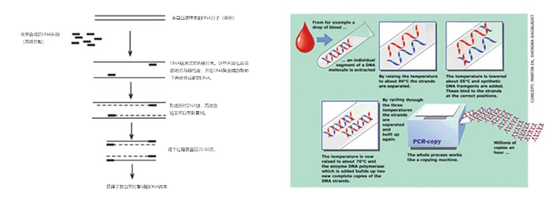
电泳 使用凝胶电泳可以根据分子大小分离DNA和RNA分子,具有适度的分辨能力。由于核酸因其磷酸基团而带负电,在中性至碱性pH值下,核酸会在电场中向阳极迁移。分辨率和迁移速度取决于凝胶中孔的直径,以及核酸的大小和结构。 最常用的基质是琼脂糖或聚丙烯酰胺。电泳可以在单链和双链的DNA上进行。在第一种情况下,DNA以长的线性链的形式通过基质迁移,迁移速度取决于大小,小分子的迁移速度比大分子快。 在双链DNA的情况下,提高温度会使DNA分子变性,如加入甲酰胺、应用极端的pH值、低离子强度的缓冲液或这些的组合。 根据变性过程中或变性后的条件,分子将采取线性、开放线圈(0~4℃)或三维(紧凑线圈)结构(图1a)。一个碱基的不同可以导致一个独特的紧凑线圈构象,这反过来又改变了电泳的流动性。 单链构象多态性试验就是基于这一原理,并在10年前被用于确定点突变。这种技术已经被高分辨率熔解曲线分析(HRMA)所取代,它基于DNA结合染料对DNA双链构象的选择性结合能力(图1b)。 图1 | 重新变性和变性条件下dsDNA的变性和重新变性。dsDNA的构型在很大程度上取决于化学和物理因素 a 高温、极端的pH值、变性物质(如甲酰胺)和低浓度的盐会破坏Watson-Crick碱基配对,导致单链分子处于开放线圈结构(ID)。在适当的条件下,原始形式的再熟化是可能的;然而,在复杂的、高分子量的DNA中,这个过程最初会运行得很慢。当开环结构在冰上被冷却时,两条链都不会再变性。在一定条件下,将形成一个具有内部碱基配对的紧凑线圈(IA),或者分子将保持在开放线圈配置(IB)。对核酸构型的操作是杂交和PCR检测的基本原则。 b 用荧光染料(如EvaGreen®)观察dsDNA的变性情况。从65℃到95℃小幅度升温,使两条DNA链之间的碱基配对逐渐解开;富含AT的区域最不稳定,首先成为单链。相应地,荧光染料也会丢失。超过一定的温度,取决于碱基的组成和DNA的大小,所有的荧光色素将回到溶液中,荧光将变得最小。当使用荧光数据和温度时,熔融曲线可以直观地显示这一过程。当50%的碱基被解离时,荧光的损失是最迅速的。这个点要用数学方法计算,叫做Tm。 其中一个应用是变性凝胶电泳(DGGE),在电泳缓冲液中加入甲酰胺/尿素混合物,dsDNA和/或部分dsRNA被变性为单链分子。这可以根据大小、电荷和构型进行分离。 1.1、电泳基质 多糖琼脂糖是从海藻中分离出来的,由酸性二糖琼脂糖的长链组成(图2)。它在室温下是一种稳定的固体粉末。 图2 | 作为分子筛的琼脂糖 a 琼脂糖由二糖琼脂糖(1,3-连接的D-b-半乳糖和1,4-连接的3,6-脱水-a-L-半乳糖)组成。 b 琼脂糖具有酸性,因为有带负电的侧基(SO4-,COO-,丙酮酸)。水合的正离子,如Na+,在暴露于电场时将迁移到负极。这样一来,带负电的核酸分子会在相反的位置迁移,而这些分子会被延缓。大分子的延迟最大。 c 琼脂糖凝胶的制备。首先,固体琼脂糖被转移到一个被加热的缓冲液中。将形成一个透明的溶液,其中所有的琼脂糖分子都以单体聚合物的形式存在。琼脂糖的凝胶化在冷却后发生,琼脂糖聚合物将组织成双螺旋。密度是由浓度决定的。事实上,琼脂糖浓度越高,基质就越紧密,分子筛就越强大。 高度纯化的形式被用于分子生物学应用。通过将其加热到56℃以上,琼脂糖在水中融化并成为水合物。冷却后,这种混合物会凝固成均匀的略带乳白色的物质。该混合物由琼脂糖聚合物组成,其密度取决于浓度。琼脂糖的浓度越高,网络就越紧密,越多的琼脂糖聚合物被组织成双α螺旋。紧密聚合的网络阻碍了核酸在琼脂糖凝胶中的扩散。琼脂糖不能用于小于40或大于约30 × 10^3个核苷酸的片段。 聚丙烯酰胺是在使用前立即由单体丙烯酰胺和交联单体双丙烯酰胺合成的。双丙烯酰胺产生的交联数量决定了链间孔隙的大小。强交联的丙烯酰胺凝胶特别适合于高分辨率的电泳,甚至可以达到1个碱基的大小(例如用于测序)。 需要注意的是,使用有神经毒性的(双)丙烯酰胺工作需要采取一定的安全预防措施,如使用手套和单独处理废物。电泳后,特定的染料可以使核酸可视化。 1.2、特殊的电泳技术 下面将介绍两种特殊的电泳技术。特别是第一种技术被经常使用。 1.2.1、毛细管凝胶电泳(CGE) 毛细管凝胶电泳(CGE)是传统凝胶电泳的一种小型化形式。在毛细管中,聚合物起着分子筛的作用。在变性条件下,复杂的DNA混合物可以以高分辨率(可以检测到一个碱基的差异)按大小进行分离,数量非常少,常用于病理学中微卫星不稳定性(MSI)的基因分型。 1.2.2、脉冲场凝胶电泳(PFGE) 为了分离非常大的基因组DNA片段(高达几Mbp)或完整的染色体,需要进行脉冲场凝胶电泳。使用非常松散的琼脂糖凝胶(约0.3%的高凝胶强度琼脂糖)和非常低的电流,可以分离大小达50kb的DNA分子。 当在凝胶中移动时,分子被置于一个电场中,其方向以特定的模式不断改变。长的DNA分子在带正电的阳极方向上迁移得非常缓慢。分子的方向与电场平行:改变电场的方向会改变DNA分子迁移的方向。 DNA分子只有在完全重新定向后才开始移动。相对较小的分子会比大分子更迅速地重塑自己,导致小片段和大片段之间的移动性存在净差异。PFGE是一个漫长的过程,可能需要2~7天。 02 杂交 杂交是基于具有互补碱基序列的分子(称为探针)对特定DNA或RNA序列(称为目标)的识别(图3)。 图3 | 变性、再变性和与标记探针杂交的原理示意图概述。 双链分子在变性后将变成单链,而高结构的单链分子将失去其内部碱基配对。在单链探针存在的情况下,探针和单链或(部分)双链目标分子之间将在重新变性条件下产生复合物。在杂交反应中通常使用过量的探针,因此探针分子的主要部分将留在溶液中 目标和探针之间16~10,000个碱基的Watson-Crick碱基配对是该技术的关键事件。探针通常是RNA或ssDNA分子,但也可以是蛋白核酸(PNA)(图4)或锁定核酸(LNA)。 图4 | PNA和DNA之间的比较 蛋白质核酸(PNA)由一个肽的骨架组成,氮基附着在这个骨架上。肽骨架的独特结构使每个碱基(A、G、C、T)之间的距离与DNA分子中的相同。PNA在稳定性和杂交强度方面比DNA或RNA有许多优势。 杂交可以发生在DNA和RNA序列上。碱基配对只有在目标物为单链且为开环结构时才有可能(见图1)。杂交有多种可能性,因为它可以在溶液中发生,使用固体载体(如尼龙膜、ELISA微孔板或玻璃),甚至使用细胞或组织中的固相。 首先,变性破坏了碱基配对的氢键,从而将原生分子的双链螺旋分离成两个单体。由于探针的小尺寸和高浓度,原生DNA链之间重新变性或RNA结构重新折叠的几率非常低,与探针的杂交发生在偏好的位置。与探针相连的标签是检测反应的后续起点。在经典的杂交中,探针被标记了。 杂交是一种技术,用于直接分析方法或作为分子诊断程序的一部分。直接分析的例子是使用荧光探针识别单个肿瘤细胞的DNA扩增,或用10^6个探针分析DNA芯片上肿瘤的所有细胞(荧光)RNA的表达。引物和/或探针的杂交是在分子分析中获得反应选择性的一种工具,如(q)PCR。 03 扩增 在过去的三十年里,人们开发了几种方法,在酶促核酸合成的帮助下,对低浓度的核酸片段进行扩增。靶标和探针都可以被扩增,但聚合酶链反应的靶标扩增几乎胜过了所有其他方法。扩增大大提高了分子诊断技术的技术特异性和敏感性。 诊断学中最相关的扩增方法是聚合酶链反应(PCR)。聚合酶链反应允许通过反复的循环,使两个特定的寡核苷酸(引物)与DNA目标杂交,形成聚合酶合成DNA的起点,从而实现双链DNA片段的指数化酶促合成。 这些序列一般相隔60~400个碱基。有几个因素很重要:材料的来源、进行的PCR类型以及PCR后产品的最终用途。在某些特殊条件下,有可能扩增出全长的cDNA。 合成从引物的3′-OH端开始向上和向下退火。这些引物提供了一个短的双链片段,被热稳定的、依赖DNA的DNA聚合酶识别。两个新的双链DNA分子(称为扩增物或扩增子)是最终产品。它们可以通过随后的加热循环变性(图5)。 图5 | 聚合酶链式反应(PCR) PCR使用一个DNA基质和一个以退火引物为起点的短双链片段。这一过程的选择性是通过引物的碱基与热变性的ssDNA靶体退火的序列实现的。DNA目标的两条链都可以作为两种不同引物的基质。合成的方向是5′-3′。一旦合成完成,新的加热(变性)将开始,引物退火和DNA合成的循环将再次开始。两个循环后将产生目标的第一个拷贝。产品的长度将由两个选定的引物在目标上的退火位置决定。每个周期从两个DNA分子开始,每个周期后将增加一倍。因此,新产品的合成具有2n功能,25-30个周期后将产生数百万个拷贝(扩增物或扩增子)。 b显示了诺贝尔委员会网站上公布的DNA扩增仪(© 诺贝尔基金会)。1993年,K. B. Mullis因这一发现获得了诺贝尔化学奖。 04 DNA序列分析 核酸鉴定的最终形式是对化学结构的阐明。这可以通过DNA序列分析或测序来详细实现。这种技术用于确定核苷酸的主要序列(碱基顺序):哪个氮基在单链核酸分子的糖-磷酸盐骨架的第1至n位,从5′-3′开始。 在过去的70年里,各种测序方法被开发出来。目前,桑格在1975年开发的Sanger 测序法的变种被用于诊断中。核苷酸的衍生物在加入后会阻碍链的进一步延伸,这是根据这种方法进行测序的基础。以前,使用的是放射性标记的核苷酸。 如今,在「染料终止器测序」中,使用的是荧光染色剂。每个双脱氧核苷酸都用自己特定的氟色素标记。由于四种不同颜色的荧光,这些可以在一个反应容器中同时检测。 05 生物物理学方法 最近,生物物理方法发展非常迅速,并被应用于传染病诊断中病原学表型的快速和准确识别。目前在微生物学领域有各种旨在鉴定的生物物理平台。这些技术之一是拉曼光谱法:一种用于识别复杂分子的通用技术。 不仅是蛋白质和核酸,而且复杂的成分,如细菌或真菌细胞的核糖体蛋白也可以被识别。在基质辅助激光解吸/电离飞行时间质谱法(MALDI-TOF MS)中,生物质被添加到一个基质(例如甲酸)中。 该基质用于促进激光对分子进行电离。然后这些被电离的成分根据其大小和电荷(m/z)被分离。离子到达一定距离的检测器所需的时间(=速度)被测量。这导致了某种峰型(光谱),可以与数据库进行比较。挑战之一是测量微生物时这些光谱的稳定性。 好了,以上就是今天分享的全部内容,如果大家有什么疑问,欢迎在后台给我们留言,或者加入我们一起讨论,若觉得文章不错,也请关注我们,以免错过后续更新~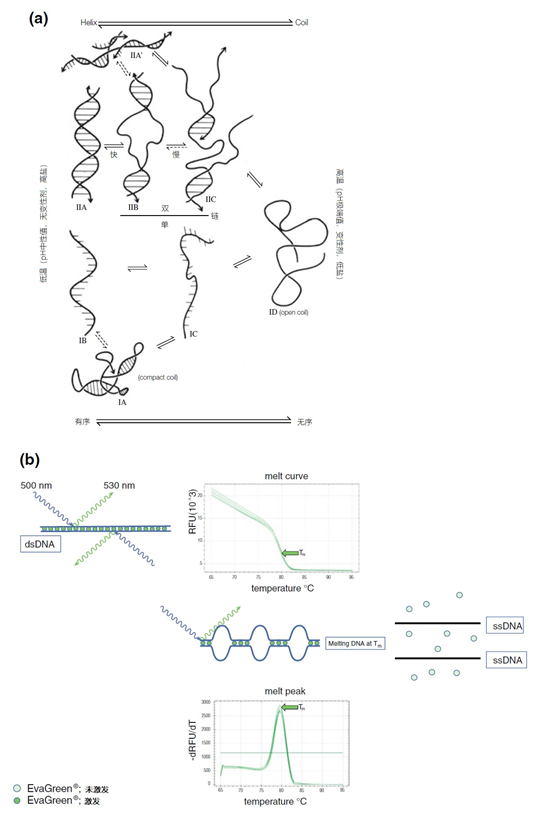

来源:诊断科学
声明:本平台注明来源的稿件均为转载,仅用于分享,不代表平台立场,如涉及版权等问题,请尽快联系我们,我们第一时间更正,谢谢!






